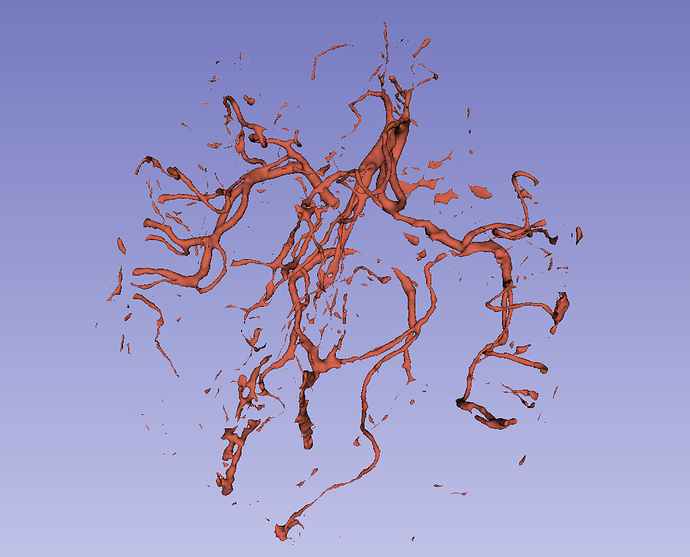西安交通大学第一附属医院神经外科 杜昌旺
安装及使用swiss skull stripper见济南军区总医院神经外科王奎重教授的教程贴 利用3D Slicer自动剥离CT或MRI影像上的头皮和颅骨
我在操作过程中强调一下可能出现的问题。
展开阅读全部
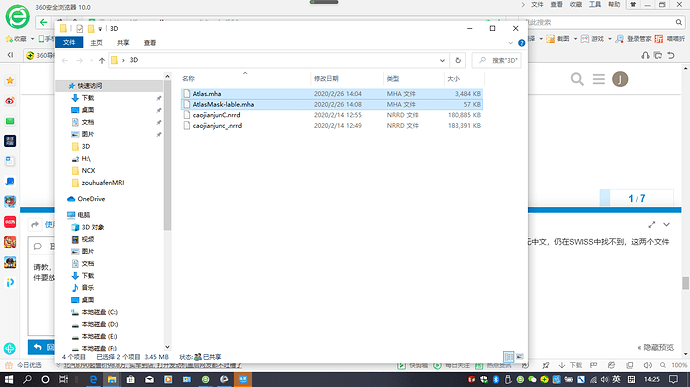
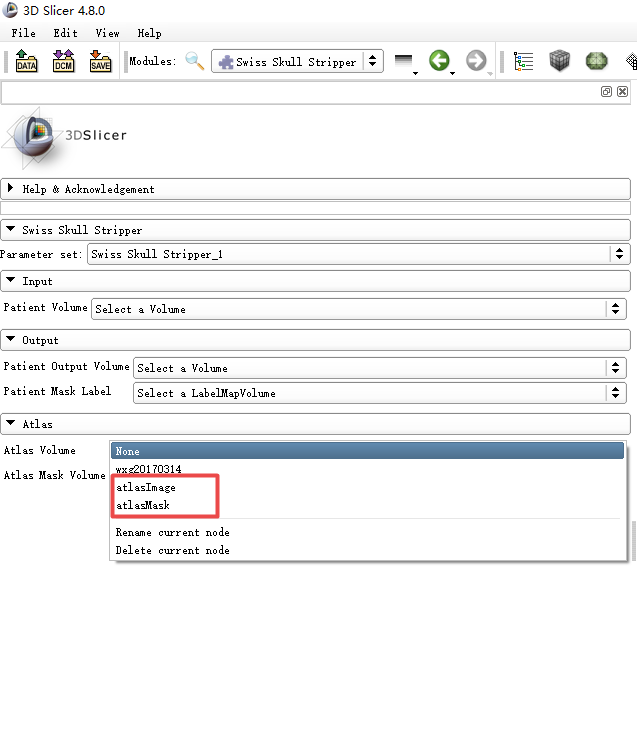
从官网上下载的这两个文件:atlas image、atlas brain mask,把它们保存在无中文名字的文件夹内待用,
![]()
并修改文件名分别为Atlas.mha(不修改或修改任意文件名Patient Out Volume可以识别)和AtlasMask-label. mha(这个文件必须修改为AtlasMask-label. mha,否则Patient Mask Label选项不能识别这个文件 )。
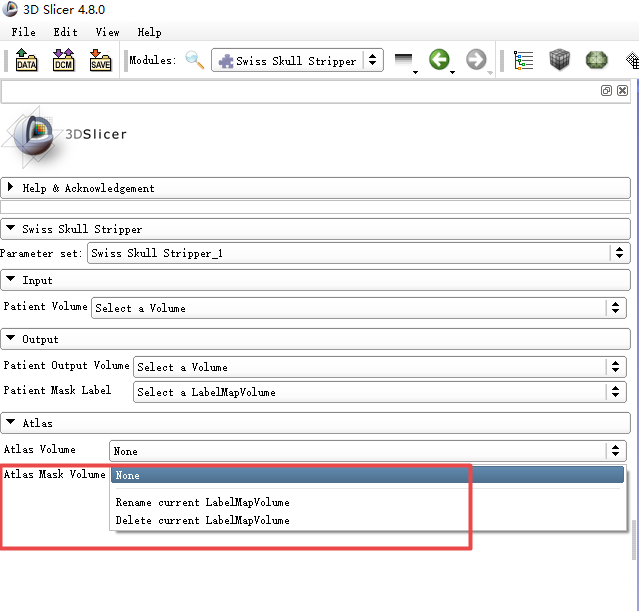
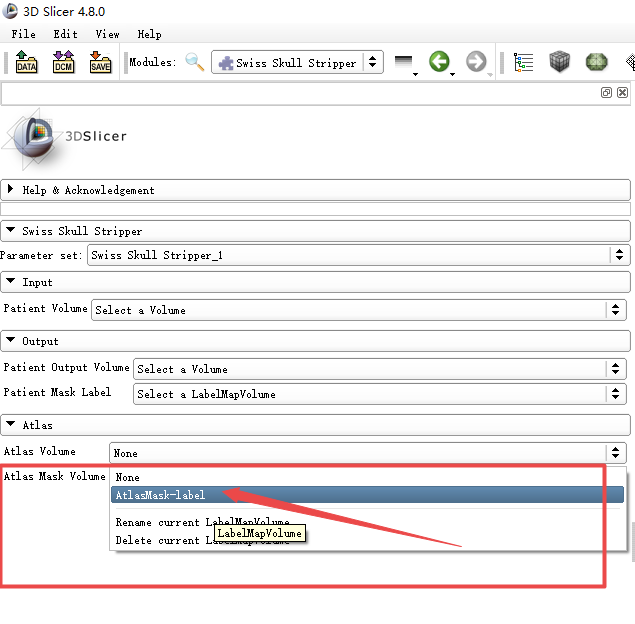
修改文件名后重新载入,看是不是能识别了,这个是关键。
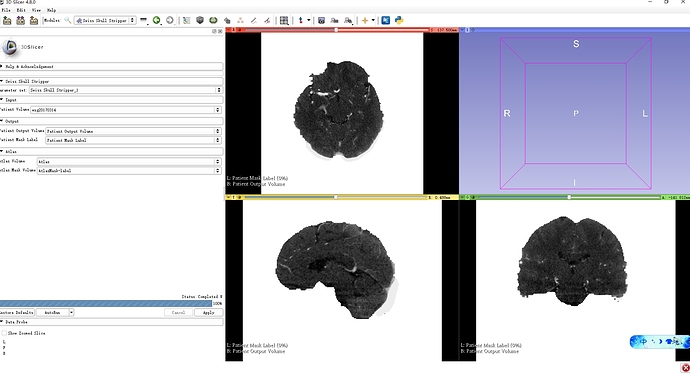
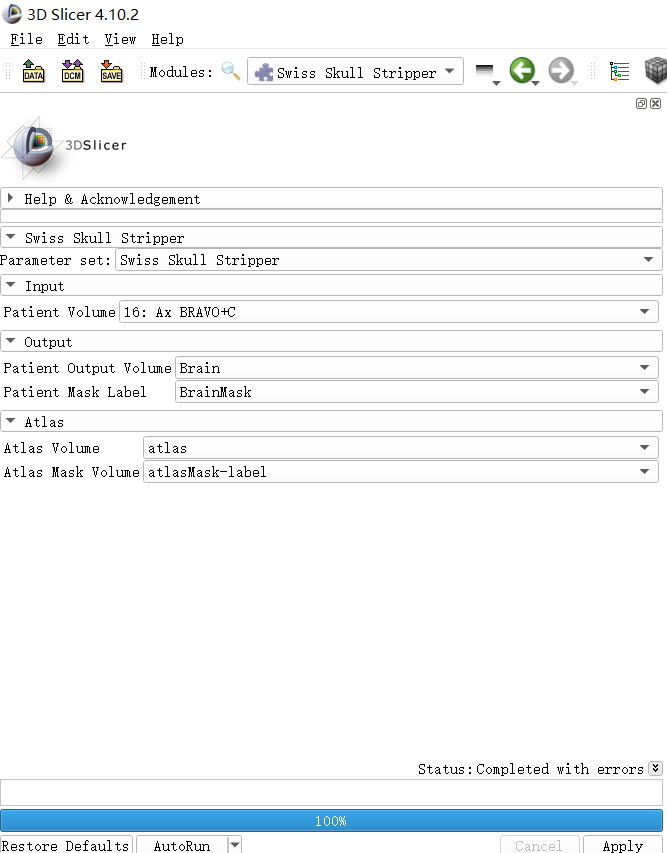
进入Swiss Skull Stripper模块,选择病人的影像系列。
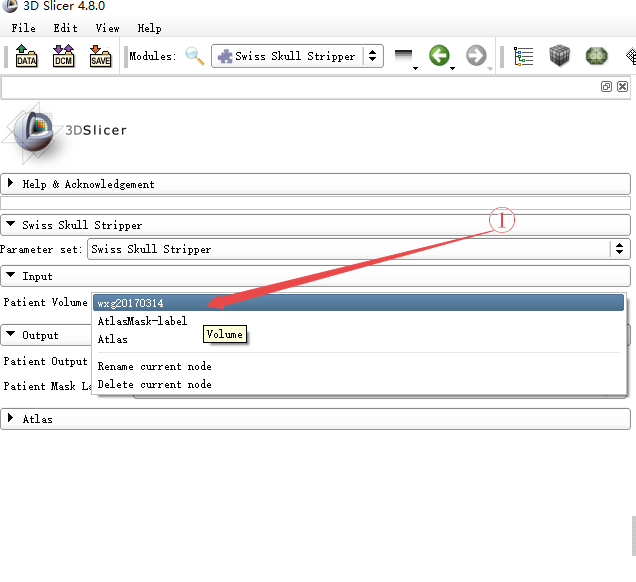
选择输出患者的Volume及Mask Label名称,选择默认即可,但是最好能做标记,不然处理多了,你自己也不知道三维建模的时候用哪个系列
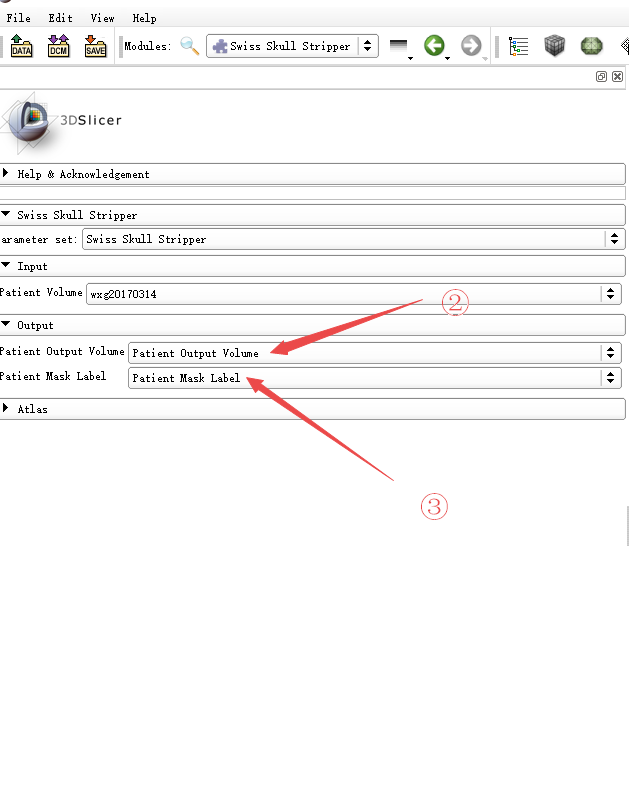
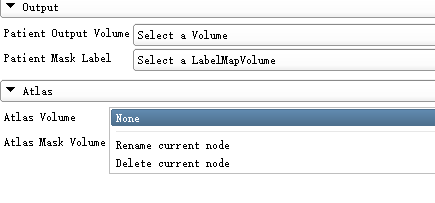
选择Atlas中刚下载更名的文件,注意不能选择错了,Atlas Volume要选Atlas,Atlas MaskVolume要选择AtlasMask-label,然后点Apply。
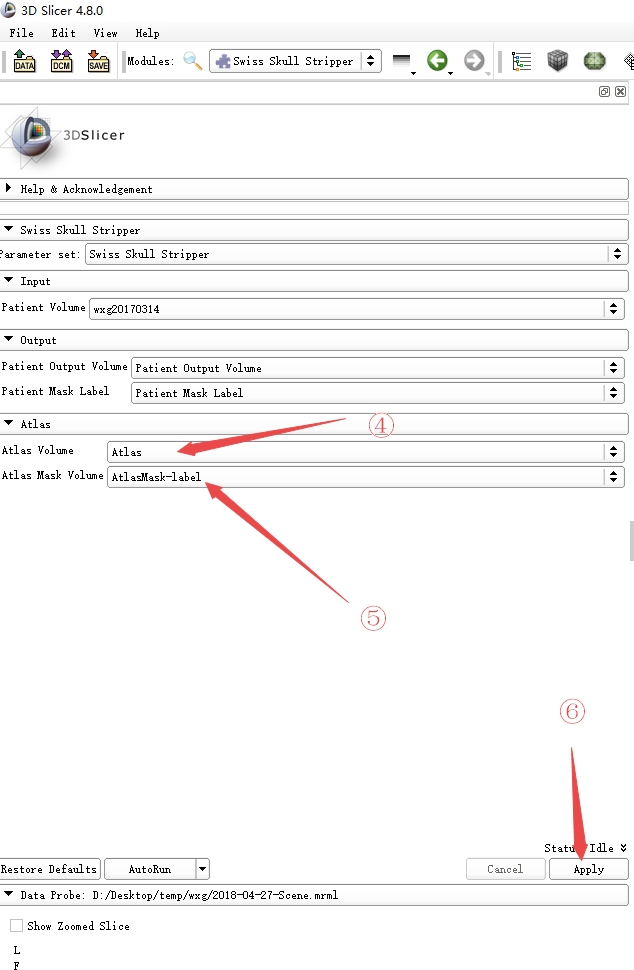
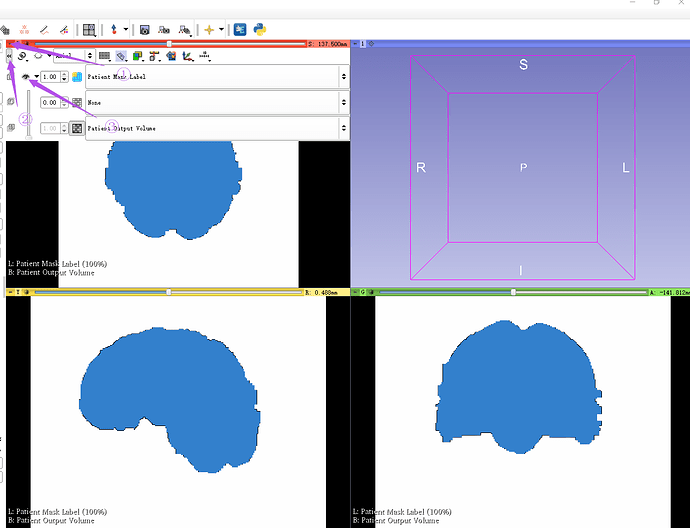
运行完了后得到如下结果
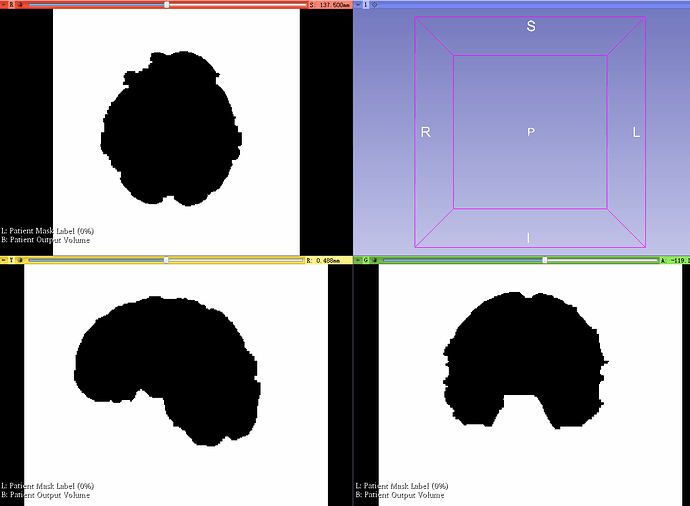
然后点击每个位像框的左上角的图钉,弹出框里点击>>,最后点击眼睛图标使其闭上,显示的就是去除头皮和颅骨的CTA像(名称为Patient Output Volume)。
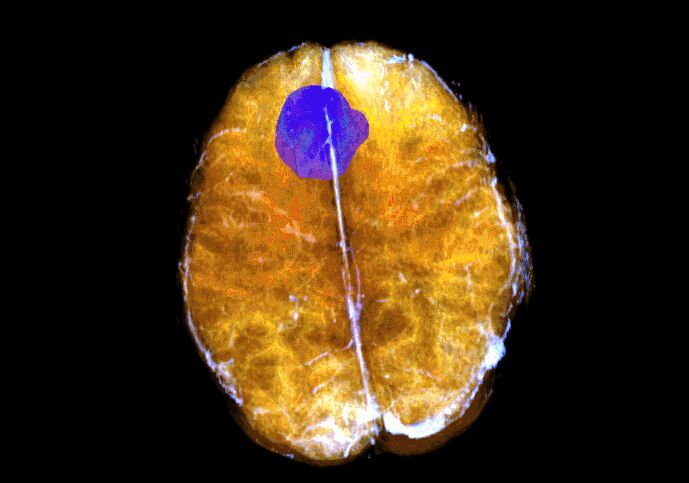
噢,天啦,咋是黑黑的图像,不像MRI剥离头皮颅骨教程里教的哪样?这也是我刚开始很久没有想明白的地方。
嘿嘿,最后弄明白了,调节窗宽窗位试试。
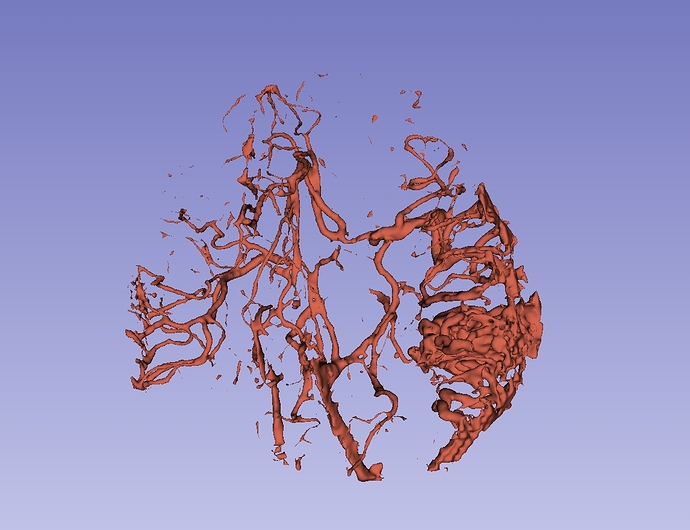
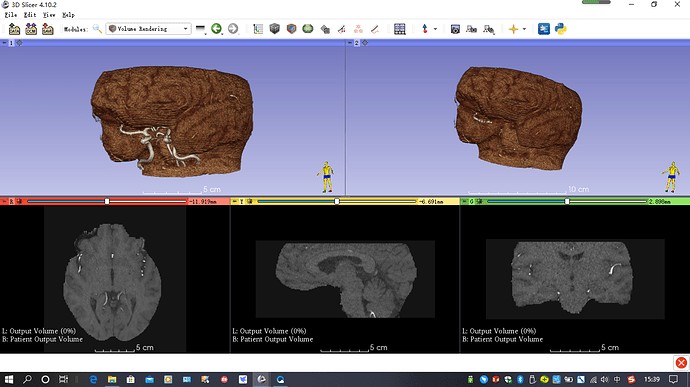
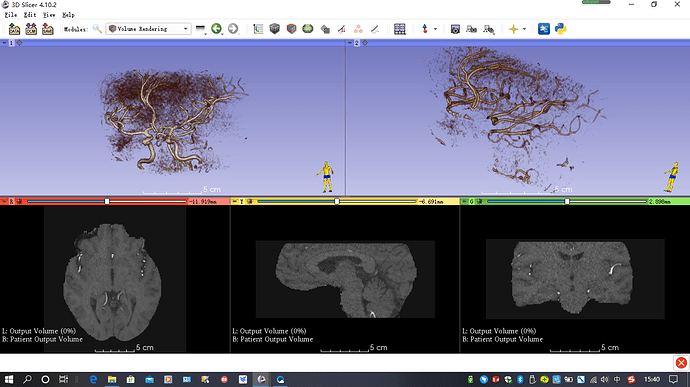
然后重建血管影!我是用segment edit编辑的。下图为术前的血管畸形的三维重建。
手术方式为开颅动静脉畸形切除术,下图为术后一年复查的CTA重建。
欢迎大家交流,微信号:dcw0922