软件版本:Slicer5.2
问题描述:导入患者核磁数据(包括平扫和增强)都提示失败,用DICOM阅片软件检验过文件可以正常查看
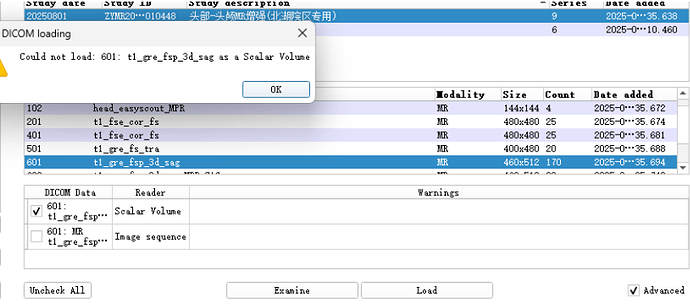
错误日志信息:Could not load: 601: t1_gre_fsp_3d_sag as a Scalar Volume
“Could not load: … as a Scalar Volume” 这个错误在 3D Slicer 社区里有不少讨论。下面是常见原因 + 排查修复建议,供你一步步尝试。
 一、错误原因可能有哪些
一、错误原因可能有哪些
根据社区经验和 Slicer 文档,出现这个错误通常是因为 DICOM 系列不能被识别为标量体 (scalar volume)。可能的原因包括:
| 原因 | 说明 |
|---|---|
| 图像不是单通道标量格式,而是 RGB / 伪彩 / 其他格式 | 如果 DICOM 的 (0028,0002) SamplesPerPixel ≠ 1,或者 PhotometricInterpretation 是 RGB、PALETTE 等,Slicer 无法当成标量图像加载。 (3D Slicer Community) |
| 位深 / 数据类型不标准 | 有些 DICOM 文件用 15-bit、浮点、压缩格式,Slicer 读不到或不支持。 (3D Slicer Community) |
| 切片间距不一致 / 采集几何不规则 | 如果切片之间的几何位置、方向、间距不一致,Slicer 可能无法把它们组织成体数据。 (slicer.readthedocs.io) |
| DICOM 标签缺失 / 损坏 / 修改过 | 比如缺少 ImagePositionPatient、SliceThickness、SeriesInstanceUID 等关键标签;或者经过匿名化 / 清洗后标签被破坏。 (slicer.readthedocs.io) |
| 文件路径 / 名称中有特殊字符 | 有报告说如果路径或文件名中含有非 ASCII 或重音符号,导入可能出错。 (3D Slicer Community) |
| DICOM 不是原始医学影像,而是仪器软件生成的二次捕获图像 | 有些设备导出的 DICOM 是截图 / 渲染形式,不是真正体数据,Slicer 识别不了。 (3D Slicer Community) |
 二、排查步骤与修复建议
二、排查步骤与修复建议
下面是按顺序操作的建议,你可以一步步尝试,看看哪个环节出问题。
| 步骤 | 操作 | 目的 / 检查点 |
|---|---|---|
| 1 | 在 Slicer 中打开 DICOM 模块 → DICOM Browser → 查看该 series 的 metadata(右键 → View Metadata) | 检查 (0028,0002) SamplesPerPixel、(0028,0004) PhotometricInterpretation、(0028,0100) BitsAllocated / BitsStored / HighBit、SliceThickness、ImagePositionPatient 等标签是否合理 |
| 2 | 用 DicomPatcher 扩展模块修补 DICOM | Slicer 社区推荐使用 DicomPatcher,可以修正一些标签或格式问题。 (3D Slicer Community) |
| 3 | 切换 DICOM 读取器(DCMTK / GDCM) | 在 Slicer 设置里有 DICOMScalarVolumePlugin 的读取器选项,尝试换一个,看是否能加载成功。 (slicer.readthedocs.io) |
| 4 | 将文件移动到路径只含 ASCII 字符、无空格 / 特殊字符的目录 | 避免路径 / 文件名造成读取失败。 (3D Slicer Community) |
| 5 | 检查是否是 RGB / 多通道图像 | 如果是 RGB(SamplesPerPixel=3),Slicer 无法当作标量体加载。可尝试转换为灰度或单通道格式。 (3D Slicer Community) |
| 6 | 使用外部 DICOM 转换工具(如 dcm2niix)将 DICOM 转为 NIfTI / NRRD,再在 Slicer 中加载 | 这种方式能绕开 DICOM loader 的限制。 Slicer 内部也有 dcm2niix 支持。 (slicer.readthedocs.io) |
| 7 | 尝试更高 / 最新版本的 Slicer | 有时新版修复了对特殊 DICOM 的兼容问题。部分用户报告在更新或使用 Preview 版本后能成功加载。 (3D Slicer Community) |
 三、一个可能适用的解决流程(示例)
三、一个可能适用的解决流程(示例)
假设你手头这组 MRI/增强扫描无法加载:
- 在 DICOM Browser 里看到该系列
t1_gre_fsp_3d_sag,右键 → “View Metadata”,注意看上面提到的关键标签; - 如果看到 SamplesPerPixel = 3 或 PhotometricInterpretation = RGB,就说明它是彩色 / 非标量图像,Slicer 默认不能加载;
- 安装 DicomPatcher 扩展,在该模块下对该系列 patch 修正;
- 或者把该序列用dcm2nii扩展模块打开;
- 如果目录 / 路径名中有中文、空格、特殊符号,改为简单 ASCII 路径再重试;
- 切换 DICOM reader(在 Preferences 或 Application Settings / DICOM / DICOMScalarVolumePlugin)尝试 DCMTK vs GDCM;
- 如果还加载不上,可考虑把该系列上传至论坛 / GitHub(去隐私化)请开发者直接查看结构。
如果你愿意,可以把那组 无法加载的 DICOM 文件的 metadata(不带隐私信息) 发给我(或贴出来关键标签),我可以帮你具体分析是哪项标签或格式不兼容。