软件版本:5.8.0
问题描述:对ROI进行勾画后,在导出时选择导出lablemap然后保存到文件,和直接导出到文件有什么区别吗?我接下来要用pyradiomics提取特征,应该选择哪个方法?
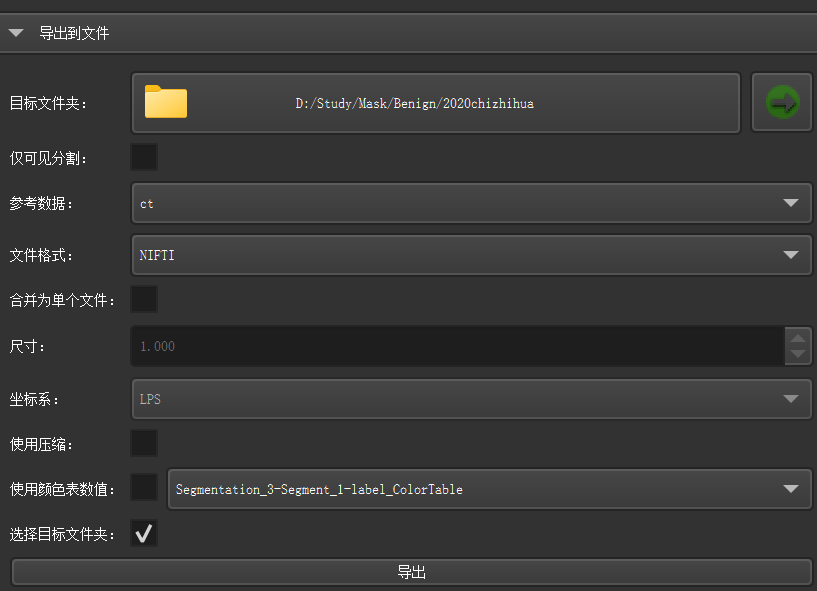
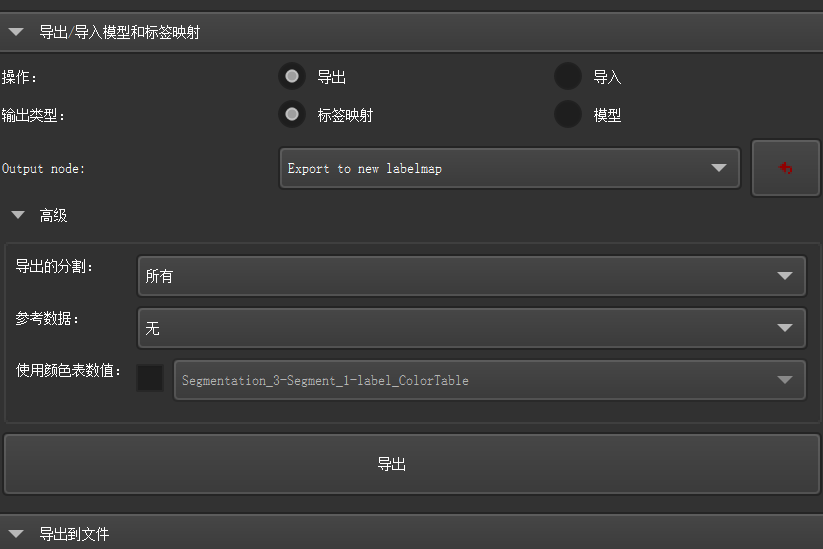
你贴的图片显示的界面来自 3D Slicer 的 “分割 Segmentations)” 模块,其实“导出到 Labelmap” 和 “直接导出到 文件” 两种方式看起来类似,但底层区别和适用场景不同,尤其在你要用 PyRadiomics 提取特征 时非常关键![]()
 一、两种导出方式的区别
一、两种导出方式的区别
| 项目 | 导出到 Labelmap | 直接导出到文件 |
|---|---|---|
| 导出位置 | 先在 Slicer 场景内生成一个新的 LabelMap 节点(即体素掩膜),之后你可以再另存为 nii/nrrd。 | 直接将分割结果写成文件(如 NIFTI、NRRD)保存到磁盘。 |
| 文件生成逻辑 | 两步:① Segmentation → LabelMap 转换;② File → Save 保存。 | 一步完成:Segmentation 自动转换后直接输出到目标目录。 |
| 控制参数 | 可指定参考体积(align、spacing)、是否高精度导出、合并段等。 | 参数较少,主要依赖当前 Segmentation 内的几何定义。 |
| 结果节点 | 会在 Data 模块里多出一个 LabelMapVolume 节点,可再处理。 |
不会在 Slicer 场景中生成额外节点,仅在磁盘生成文件。 |
 二、对于 PyRadiomics 的正确输出格式
二、对于 PyRadiomics 的正确输出格式
PyRadiomics 的输入通常是:
-
原始影像(CT/MRI) →
.nii或.nrrd -
对应的掩膜(ROI) → 二值 LabelMap
.nii或.nrrd
![]() 推荐导出路径:
推荐导出路径:
先“导出到 Labelmap” → 然后保存 LabelMap 为
.nii.gz文件
理由如下:
-
PyRadiomics 需要掩膜与影像在相同空间、像素间距、方向上严格对齐。
“导出到 Labelmap” 时可以设置参考体积(Reference volume)来保证这一点。 -
直接“导出到文件” 有时不会自动应用参考影像对齐,可能导致掩膜与原图空间错位(尤其多段 ROI 时)。
-
LabelMap 文件是标准的整型标签图(如 0=背景, 1=ROI),正是 PyRadiomics 所要求的格式。
 三、推荐操作步骤
三、推荐操作步骤
-
在 Segment Editor 中完成 ROI 勾画;
-
打开 Segmentations 模块 → Export/Import models and labelmaps;
-
选择:
-
操作:导出
-
输出类型:标签映射(labelmap)
-
参考数据:选择原始 CT/MRI;
-
-
点击 “导出”;
-
在 Data 模块中右键新生成的 LabelMap → “另存为 (Save)”;
-
保存为
.nii.gz或.nrrd; -
将该文件与原始影像文件一起输入到 PyRadiomics。
 四、补充建议
四、补充建议
-
若你有多段 ROI(如膀胱壁、腔内、病灶),PyRadiomics 可设置
label参数指定提取哪一段。 -
导出时若想把所有段合成单一掩膜,可在导出界面勾选 “合并为单个文件”。
-
记得保持 “Use reference geometry” 选中,以确保坐标一致。
![]() 结论:
结论:
对于 PyRadiomics 特征提取,请选择“导出到 Labelmap”再保存为 nii(推荐
.nii.gz)。
“直接导出到文件” 虽方便,但容易出现空间不匹配问题,不建议用于定量分析。