软件版本:5.10.0
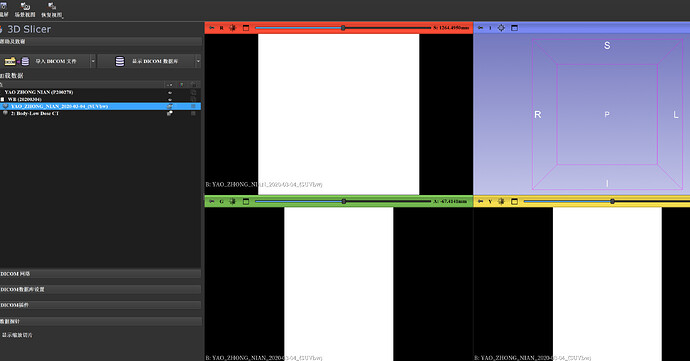
问题描述:为什么导入PET的SUVbw图像之后出现全白的显示 ![]()
![]()
先别慌,这种“全白”的情况 90% 是显示参数的问题,PET 的 SUVbw 数据本身一般是没坏的。你可以按下面顺序排查一下:
一、先确认是不是窗宽窗位的问题
-
在绿色视图里按住鼠标右键左右/上下拖动
- 左右:调窗位
- 上下:调窗宽
如果拖一拖能把图像“拉”出来(出现灰度差、轮廓),说明只是窗宽窗位不合适。
-
用 Volumes 模块重置显示
-
切到模块:
体数据(Volumes) -
在左侧下拉框里选中这组 PET:
YAO_ZHONG_NIAN_2020-03-04_(SUVbw) -
在 Display(显示)部分:
- 勾选或点击一次 Auto window/level(自动窗宽/窗位)
- 看下面的 Scalar range(标量范围),例如常见是
0 ~ 10或0 ~ 20。
-
如果图像仍然很白,可以手动设一个范围:
- Level ≈ 2.5–3
- Window ≈ 5–6(或者 SUV 最大值的一半)
-
-
换一个查找表试试
- 在 Display 里把 “Lookup table / Color table” 改成
PET、HotIron或其它伪彩色,看是否能看到代谢分布。
- 在 Display 里把 “Lookup table / Color table” 改成
二、检查数据本身是否正常
-
看 DataProbe(状态栏)里的数值
底部状态栏(或 DataProbe 模块)把鼠标放在白色区域上,看当前像素值:- 如果是 0–10 之间浮点数(比如 1.8、3.5),数据是正常的,只是显示设置问题。
- 如果整幅图扫来扫去几乎都是同一个值(比如全是 0 或一个固定大数),那可能是这组 SUVbw DICOM 导出有问题。
-
对比一下同一病人的原始 PET 或 CT
- 在 DICOM 浏览器里,把 原始 PT 图像 和 Low Dose CT 一起加载,看 CT 是否正常显示;
- 若 CT 正常、PET 原始图像正常,只有 SUVbw 这组异常,全白问题多半来自 SUVbw 序列本身的编码或导出。
三、如果确实是 SUVbw 序列本身有问题
如果按照上面的办法:
- 窗宽窗位调整无效,
- Scalar range 显示为奇怪的值(如整个 volume 都是同一个值),
建议:
-
在医院工作站上先看一眼这组 SUVbw
- 若在工作站上也显示异常,重新导出:优先导出原始 PET 图像(Counts)+ CT,后续在 Slicer 里再做 SUV 计算或仅用原始 PET 显示。
-
重新导出 DICOM
- 导出时尽量选择“原始 PET 图像 + CT”,不要只导出后处理的 SUVbw 影像;
- 有条件的话,可以导出 NIfTI 或其他格式试一次。
四、一个实用的小建议
做 PET/CT 的时候,可以采用下面的流程,基本不会踩坑:
-
从工作站导出:Low-dose CT + 原始 PET(不强求 SUVbw)。
-
在 Slicer 中:
- CT 用常规骨窗/脑窗;
- PET 用 Volumes 模块调整窗宽窗位,或挂伪彩色。
-
如果确实需要 SUV 值,后续可以用专门的 PET 插件或在工作站上量化,但常规阅片和配准在 Slicer 里足够了。
你可以先按“右键拖动 + Volumes 模块自动窗宽/窗位 + 看 scalar range”这三步试一下,如果仍然是纯白,截一个 Volumes 模块的界面(包含 Scalar range 和窗口设置)给我,我帮你再具体判断是显示问题还是 SUVbw 数据本身出了问题。