旋转数据转换成切片数据的过程只能在血管机器的工作站中完成,目前slicer还不具备相应的模块及功能,我已经咨询了软件开发者。
DSA数据的重建必须有三维旋转的数据才能重建么?
对呀,3D旋转数据的轴位图像
好的,了解,感谢了!
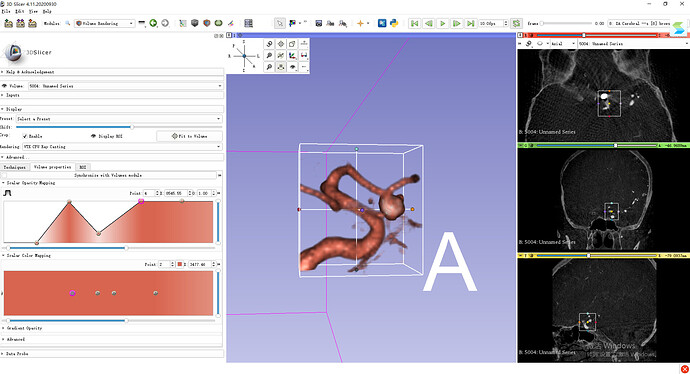
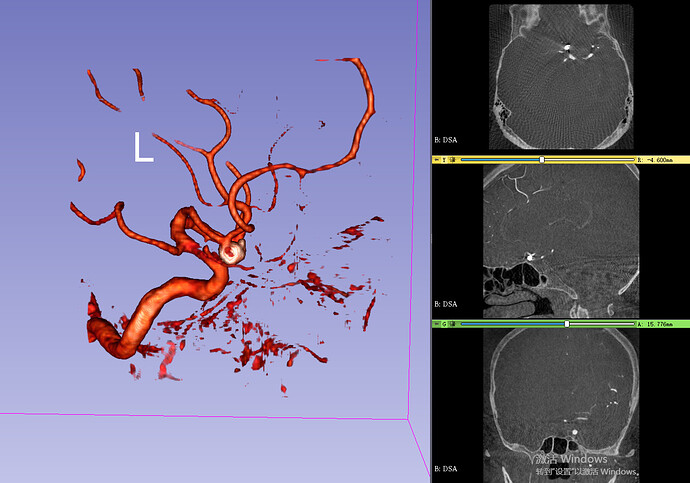
各位老师,应用DSA数据重建出来的血管床突段显示比较清楚,但是周边还是有一些杂质的影像,在Volume rendering中如何让血管更漂亮?(以下为前交通动脉瘤栓塞术后病例)
我用的volume rendering重建血管发现血管周边总是有很多杂志,ROI、shift、volume properties中调节之后也达不到您所呈现图片的效果。
不知你有没有使用mango处理过原始数据,我个人经验,处理后,会好很多。
新的高配机器有切层输出,低配版的恐怕没有,其实10多年前的飞利浦3D工作站都可以直接输出3D,其格式的VRML,以前的导出方法在研究论文里面有,时间久了具体怎么操作现在记不得了。
我导出的数据是DICOM格式的数据,直接用的slicer处理。下次试试用mango转换一下试试。DICOM转换为nii.gz,然后进入slicer转换为DICOM,又再次进入mango转换为nii.gz。到此步骤就可以用了吗?