Caoyufu
(曹玉福)
1
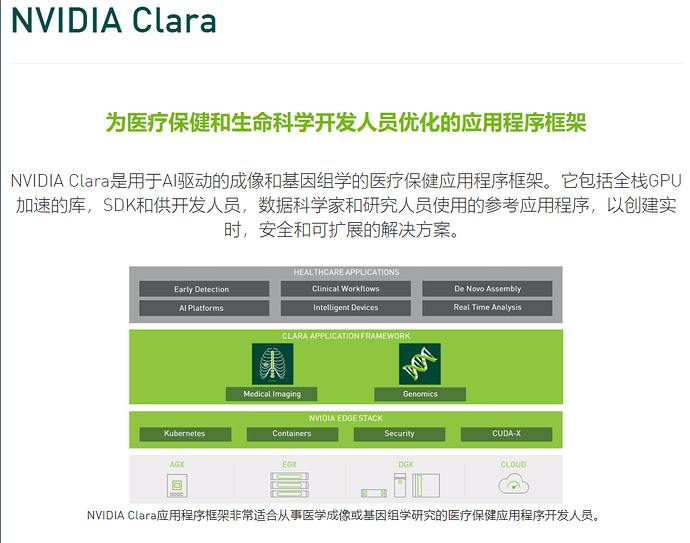
3DSlicer官方宣布,Nvidia AI辅助的分段扩展 已经可以在最新的Slicer预览版(rev28686或更高版本)中使用。该扩展由Nvidia开发,并由Slicer核心开发人员提供。虽然Slicer中还有其他AI辅助分割模块(例如DeepInfer ,TOMAAT ,SlicerCIP ),但最新添加的功能是使用Nvidia Clara ,这是一个具有强大的工业支持和对研究人员足够开放的工具包。
随着医学影像深度学习研究的兴起,正在开发更加有效和改进的方法,以通过支持GPU的模式实现AI辅助工作流程。借助最新版的NVIDIA Clara应用程序框架,开发人员拥有进行AI开发以及通过嵌入式硬件将AI功能引入医疗设备的必要工具和API。
下面视频显示了MRI脑肿瘤和肝脏分割以及全自动肝,肿瘤和脾脏分割:
教程和详细说明:请参阅Nvidia AIAA扩展文档 。
它是如何工作的?
输入的图像(和输入点-在进行引导分割的情况下)被发送到装有Nvidia GPU的计算机,该计算机运行Linux操作系统和Nvidia Clara软件 。服务器使用选定的AI模型计算细分,并将结果发送回Slicer进行显示和进一步处理。
官方在PerkLab (加拿大皇后大学) 设置了一个演示服务器, 以使Slicer用户更容易入门,而无需设置自己的处理计算机。我们上传了Nvidia开发的几个AI模型。我们按原样提供这些模型和处理服务,我们不保证此服务的质量(分段结果的有效性,速度,服务器正常运行时间等)。没有患者信息会发送到处理服务器,并且处理后图像和结果将从服务器中删除,但是用户在使用我们的服务器时需要确保遵守其数据管理准则。如果存在任何保密问题,则可以使用公开可用的图像进行测试:请参阅Slicer的样本数据模块或TCIA浏览器 扩展程序,或从其他网站下载,例如Medical Decathlon。
如果有人想分享他们的AI模型进行细分 ,只要该模型与Nvidia Clara兼容,就应该能够在服务器上安装该模型并将其提供给Slicer社区。
Caoyufu
(曹玉福)
2
Nvidia AI辅助分割可在[3D Slicer中使用],这是一种流行的免费开放源代码医学图像可视化和分析应用程序。该工具在应用程序的“细分编辑器”模块中可用。
该工具有两种模式:
- 全自动分割:无需用户输入。在“自动分割”部分中,选择模型,然后单击“开始”。分割过程可能需要几分钟(对于网络上传速度较慢的计算机上的大型数据集,此过程可能需要大约5-10分钟)。
- 基于边界点的分割:要求用户在感兴趣的结构的边缘附近指定输入点,每侧一个。细分通常需要不到一分钟的时间。
自动分割的示例结果:
设定
- 从http://download.slicer.org/下载并安装最新的3D Slicer Preview版本(4.11.x)。
- 启动3D Slicer,然后打开扩展管理器
- 安装NvidiaAIAssistedAnnotation扩展(在“分割”类别中),等待安装完成,然后单击“重新启动”
- 可选:在菜单中设置注释服务器设置:编辑/应用程序设置/ NVidia。如果服务器地址为空,则将使用默认的公共服务器。无法保证服务器的可用性或所提供型号的质量。
要设置自己的分割服务器,请按照以下说明进行操作。
对于运行预先训练的模型,具有强大的NVidia GPU的中型台式计算机(例如,具有8GB内存和RTX 2070 GPU的老式计算机)就足够了。服务器上需要Linux操作系统。
运行3D Slicer的计算机对运行AI辅助分割没有任何特殊要求。对于大型数据集,建议使用快速的网络上传速度,因为分割通常只需几秒钟,而图像数据传输只需几分钟。
教程和范例
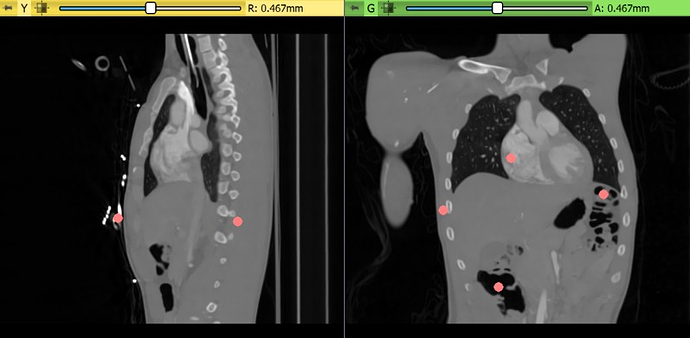
基于边界点的脑肿瘤MRI分割:
- 转到 示例数据 模块并加载“ MRBrainTumor1”数据集
- 转到 Segment Editor
- 创建一个新的Segment
- 点击“ Nvidia AIAA”工具
- 在“从边界点分割”部分中,选择“ annotation_mri_brain_tumors_t1ce_tc”(经过训练可在对比增强脑MRI上分割肿瘤的模型)
- 单击“放置标记点”按钮,然后在切片视图中的所有6个侧面的肿瘤边缘附近单击,然后单击“开始”(如果显示有关将图像数据发送到远程服务器的弹出窗口,请单击“确定”)
- 在大约10秒内,自动分割结果将出现在切片视图中。要以3D形式显示结果,请点击细分列表上方的“显示3D”按钮。要以3D显示切片图像,请单击切片视图左上角的“图钉”图标,然后单击“眼睛”图标。
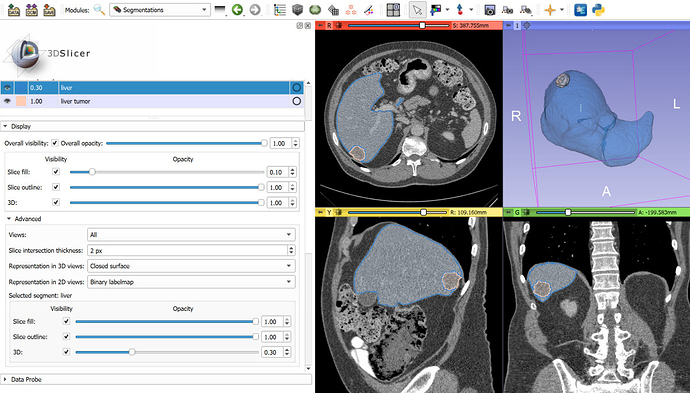
CT对肝脏的基于边界点的分割:
- 转到 示例数据 模块并加载“ CTACardio”数据集
- 转到 Segment Editor
- 创建一个新的细分
- 双击segment名称并输入“ liver”以指定细分内容
- 单击“ Nvidia AIAA”效果,“从边界点分割”部分
- 可选:单击“过滤器”图标以仅列出名称中包含“肝脏”的那些型号
- 选择“ annotation_ct_liver”(训练为在门静脉期CT图像中分割肝脏的模型)
- 单击“放置标记点”按钮,然后在切片视图中的所有6个侧面的肝脏边缘附近单击,然后单击“开始”
- 在大约30秒内,自动分割结果将出现在切片视图中。要以3D形式显示结果,请点击Segment Editor列表上方的“显示3D”按钮。
- 要在分割完成后调整先前放置的边界点,请单击“开始”按钮旁边的“编辑”图标。
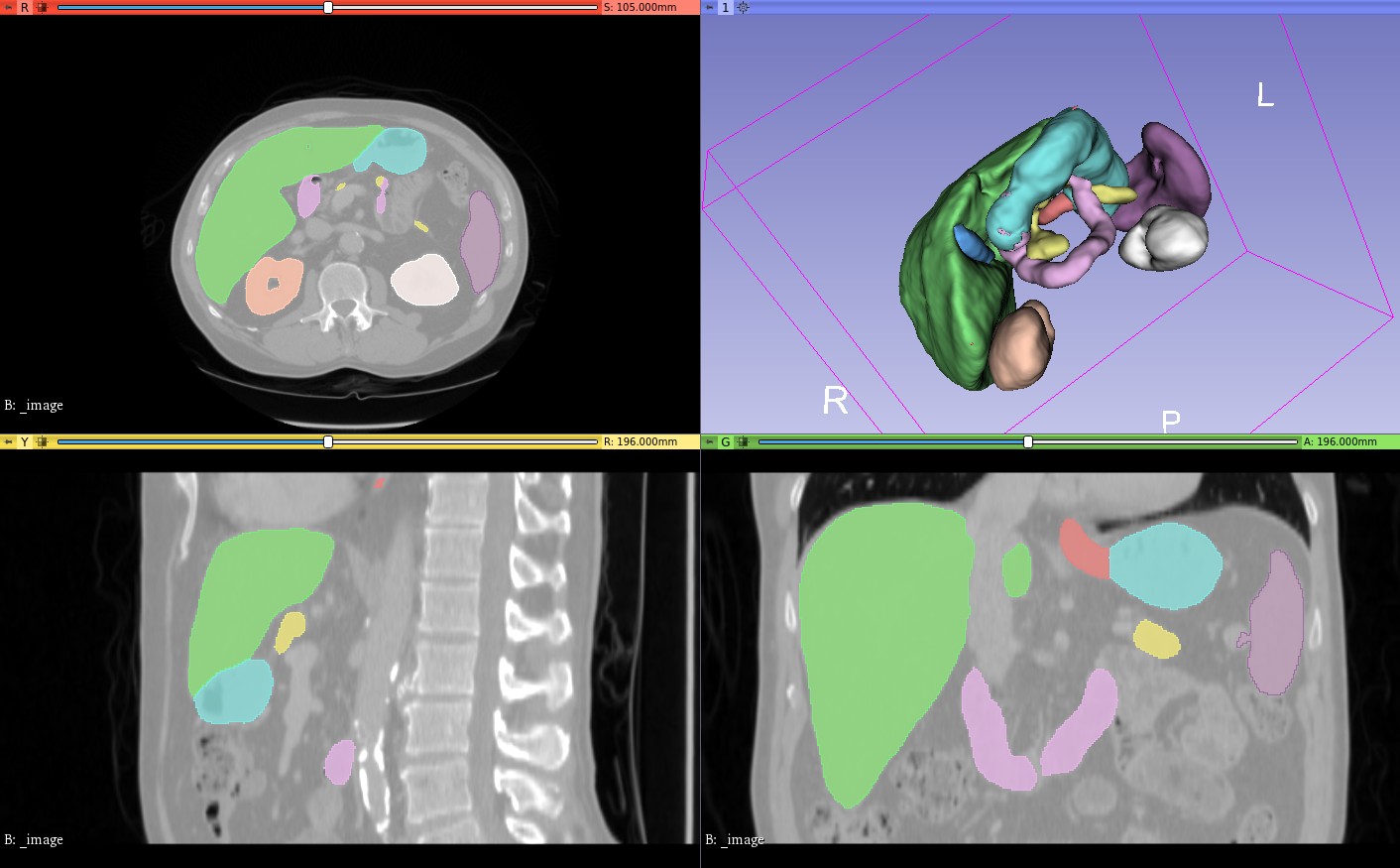
在CT上对肝脏和肿瘤进行全自动分割:
- 从http://medicaldecathlon.com/下载Task03_Liver \ imagesTr \ liver_102.nii.gz数据集并将其加载到3D Slicer中
- 转到 Segment Editor
- 点击“ Nvidia AIAA”效果
- 在“自动分割”部分中,选择“ segmentation_ct_liver_and_tumor”模型,然后单击“开始”
- 自动分割结果应在3-5分钟内显示
- 可选:转到 Segments 模块以编辑显示设置
高级
- 对于上载速度较快的本地设置服务器或计算机,请在“应用程序设置” /“ NVidia”中禁用压缩(因为压缩所花费的时间可能比传输较少数据所节省的时间要长)
- 要根据标签上的标签过滤模型,请在Nvidia AIAA效果用户界面的“模型”部分中设置标签文本。
对于开发人员
该插件可以直接从GitHub下载并安装:
- git clone https://github.com/NVIDIA/ai-assisted-annotation-client.git
- 打开3D Slicer:转到编辑->应用程序设置->模块->其他模块路径
- 添加新的模块路径:<FULL_PATH> / slicer-plugin / NvidiaAIAA
- 重新开始
- 要构建扩展包,请构建3D Slicer,然后使用CMake配置AI辅助注释客户端项目,并定义以下变量:-DSlicer_DIR:PATH = … -DNvidiaAIAssistedAnnotation_BUILD_SLICER_EXTENSION:BOOL = ON
Caoyufu
(曹玉福)
3
Caoyufu
(曹玉福)
4
1 个赞