Originally published at: http://www.slicercn.com/?p=4281
济南军区总医院神经外科 王奎重 博士
3D Slicer有强大的配准融合功能,既往推出的General Registration (BRAINS)模块虽然配准精度高,但设置参数较多,过程复杂,初学者难以掌握。本文介绍另外一个配准模块Elastix,可以对CT和MRI图像一键配准。
1.下载并打开3D Slicer4.8.1软件,安装扩展模块Elastix。关闭并重新打开3D Slicer,载入CT(6 1)和MRI(Ax 3D T1BRAVO)图像数据。在前景和背景窗口中可以看到CT和MRI图像未重叠。
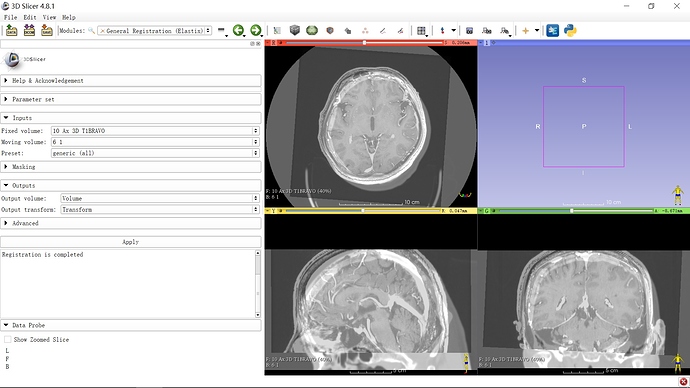
2.打开registration-general registration(Elastix)模块,在fixed volume栏里选择磁共振数据(Ax 3D T1BRAVO),moving volume栏内选择CT数据(6 1),设置输出volume和transform名字,其余为默认选项。点击Apply。
3.稍等片刻,即可配准成功。在窗口前景中选择磁共振数据(Ax 3D T1BRAVO),背景中选择新输出的数据volume,调节滑动条透明度,即可显示CT和MRI图像配准效果。
4.最后别忘了保存,可见新生成两个mha文件存在其默认文件夹中,时间长了容易变成垃圾文件,可以更改路径存入想要的文件夹。
5.若要进行图像融合,可以打开Add Scalar Volumes模块,输入磁共振(Ax 3D T1BRAVO)和数据volume,设置新融合的数据名字,点击Apply即可融合在一起。
图像配准和融合是两个不同的概念,配准是两张图像的空间位置改变使其重叠,但还是各自原来的图像。融合是两张图像融为一体不能分开,有关融合方法我以前有过介绍,这里不再赘述,实际应用中只需要配准功能就足够了。本文介绍了利用3D Slicer对CT和MRI图像一键配准融合,可以快速实现同一患者不同模式图像的融合,操作过程中也可以调节不同参数及选择不同的预设模板(preset)。笔者在实际应用中发现不同机器扫描图像通过3D Slicer打开时甚至不能在一个视野中,这可以通过粗略变换图像坐标实现;即便同一个病人扫描的MRI数据,由于扫描时间长不同序列间还是有细微的位置改变,而通过3D Slicer配准功能,可以消除这些肉眼所见到的位置改变。
注:本文仅供个人学习交流,不妥之处恳请大家批评指正。未经本人同意不得擅自转载及他用,有关参照本文产生的纠纷及法律后果本人概不负责。